 |
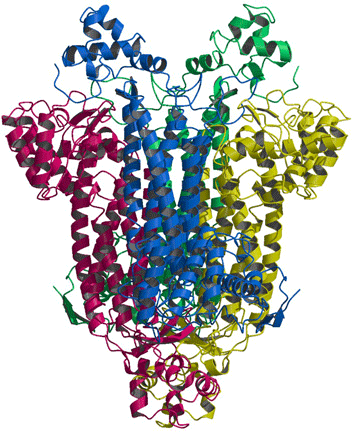
| Bacillus sp. YM55-1由来アスパルターゼの四量体分子構造 |
| 色分けされた4つのサブユニットが会合して1つの分子を形成している |
 |
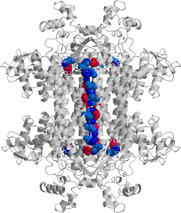
| Bacillus sp. YM55-1由来アスパルターゼの四量体分子構造 |
| 色分けされた4つのサブユニットが会合して1つの分子を形成している |
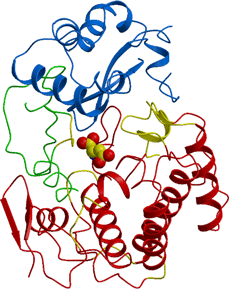 |
| 活性部位クレフトと基質結合推定モデル |
|
3つのサブユニット(赤・青・緑)により構成される活性部位クレフトを表示している。
類縁酵素との構造比較により、局所的に構造の異なる領域(黄色で表示)を見いだした。 各酵素に特徴的な機能を照らし合わせて詳細な検討を行い、基質分子であるアスパラギン酸の推定結合モデル(中央のCPK表示モデル)を作成した。 |
| イオンペアーの分布比較 | ||
| 大腸菌由来酵素 | Bacillus YM55-1由来酵素 | |
| 分子全体でイオンペアーに関与している残基 | 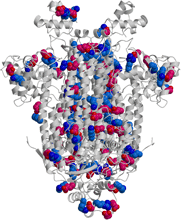 139残基 |
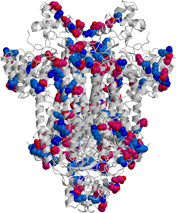 180残基 |
| ドメイン間のイオンペアーに関与している残基 | 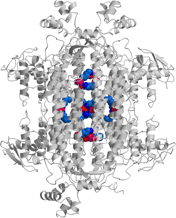 28残基 |
 48残基 |
| 全イオン性残基数 | 400残基 | 404残基 |